Estudo descreve novo mecanismo de reconhecimento de DNA viral
 Em estudo coordenado pelo Professor João T. Marques, do Laboratório de RNA de Interferência da Universidade Federal de Minas Gerais (UFMG), com a participação do professor do programa de Genética e Biologia Molecular, Dr. Eric Aguiar (Uesc), e Dr. Roenick Olmo (CNRS-França), Dr.ª Juliana Alves (ICB/UFMG), além de pesquisadores da Université de Strasbourg (França) e da Northwestern University (Estados Unidos), pesquisadores descreveram como células de insetos reconhecem moléculas de DNA viral para montar uma resposta antiviral.
Em estudo coordenado pelo Professor João T. Marques, do Laboratório de RNA de Interferência da Universidade Federal de Minas Gerais (UFMG), com a participação do professor do programa de Genética e Biologia Molecular, Dr. Eric Aguiar (Uesc), e Dr. Roenick Olmo (CNRS-França), Dr.ª Juliana Alves (ICB/UFMG), além de pesquisadores da Université de Strasbourg (França) e da Northwestern University (Estados Unidos), pesquisadores descreveram como células de insetos reconhecem moléculas de DNA viral para montar uma resposta antiviral.
Os vírus são parasitas intracelulares obrigatórios que sequestram a maquinaria da célula hospedeira. As células hospedeiras precisam reconhecer rapidamente a infecção e iniciar as defesas antivirais efetoras para evitar a replicação viral.
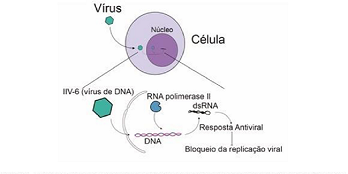
Para entender como as células infectadas reconhecem a infecção por vírus de DNA e como ocorre a sinalização para ativar respostas efetoras para controlar a replicação viral, foram utilizados como modelo a mosca da fruta (Drosophila melanogaster) e o vírus de DNA de dupla fita Invertebrate iridescent virus 6 (IIV-6), com o auxílio de diversas técnicas de Sequenciamento de Nova Geração, bioinformática, microscopia e de biologia molecular.
Nesse trabalho foi observado que durante a exposição a moléculas de DNA viral, a enzima celular RNA Polimerase II reconhece o DNA invasor e inicia sua transcrição, produzindo RNAs de fita dupla (dsRNAs), que são moléculas intermediárias que iniciam a resposta antiviral mediada pela via de RNA de interferência, que bloqueia a multiplicação do vírus.
“A descrição desse mecanismo é importante porque, de uma perspectiva evolucionária, pode nos ajudar a entender como diferentes organismos encontraram soluções distintas para o mesmo problema. Nesse caso, o mecanismo descrito é um exemplo de convergência evolutiva, pois se assemelha muito ao que observamos em células humanas infectadas por vírus de DNA”, relata o Prof. João Marques.
Segundo o Prof. Dr. Eric Aguiar, que liderou as análises de bioinformática do trabalho, “o uso do Sequenciamento de Nova Geração e Análises de Bioinformática foram fundamentais para entender os mecanismos de reconhecimento desse DNA invasor junto a possíveis mecanismos de resposta do hospedeiro para levantar hipóteses que pudessem ser experimentalmente respondidas”.
Estudos como esse são fundamentais devido a sua relevância para diversas áreas, desde a virologia e imunologia até a biotecnologia. “Os vírus de DNA são importantes patógenos de diversos organismos, como o bicho da seda, além de serem utilizados como controle biológico para alguns tipos de pragas, como é o caso da lagarta da soja”, lembra o Dr. Isaque Faria, autor do manuscrito.
Os autores ainda destacam que os achados desse trabalho, ao serem extrapolados para outros organismos, poderiam auxiliar no melhor entendimento das relações vírus-hospedeiro e contribuir para o desenvolvimento de novas estratégias de combate a infecções causadas por vírus.
O artigo foi publicado recentemente na importante revista Cell Reports, com o DOI: https://doi.org/10.1016/j.celrep.2022.110976.






















